Ejemplo de análisis espacial: riqueza de especies#
Introducción#
La riqueza de especies es el número total de especies que se encuentran en un hábitat, ecosistema, paisaje, área o región determinada.
En este cuaderno de notas se muestra la riqueza de especies de félidos (familia Felidae) en áreas de conservación de Costa Rica.
Carga de bibliotecas#
# Carga de geopandas con el alias gdp
import geopandas as gpd
# Otras bibliotecas
# Carga de pandas con el alias pd
import pandas as pd
# Carga del módulo pyplot de matplotlib con el alias plt
import matplotlib.pyplot as plt
# Carga de la clase WebFeatureService del módulo wfs de owslib
# Permite interactuar con servicios web geoespaciales tipo WFS
from owslib.wfs import WebFeatureService
# Carga de la clase BytesIO del módulo estándar io
# Permite crear un objeto en memoria que actúa como un archivo binario
from io import BytesIO
# Carga de la biblioteca Folium, para mapas interactivos
import folium
Carga de datos#
# Crear un dataframe con registros de presencia de félidos
felidos_df = pd.read_csv(
'https://raw.githubusercontent.com/gf0657-programacionsig/2024-ii/refs/heads/main/datos/gbif/gbif-felidos-cri.csv',
sep='\t'
)
# Reducción de columnas
felidos_df = felidos_df[['species', 'decimalLongitude', 'decimalLatitude']]
# Crear un geodataframe a partir del dataframe
felidos_gdf = gpd.GeoDataFrame(
felidos_df,
geometry=gpd.points_from_xy(felidos_df.decimalLongitude, felidos_df.decimalLatitude),
crs='EPSG:4326'
)
# Mostrar los primeros registros del geodataframe
felidos_gdf.head()
| species | decimalLongitude | decimalLatitude | geometry | |
|---|---|---|---|---|
| 0 | Leopardus pardalis | -83.530469 | 10.595040 | POINT (-83.53047 10.59504) |
| 1 | Panthera onca | -84.000000 | 9.950000 | POINT (-84 9.95) |
| 2 | Panthera onca | -85.050000 | 10.968080 | POINT (-85.05 10.96808) |
| 3 | Leopardus wiedii | -85.624341 | 10.370696 | POINT (-85.62434 10.3707) |
| 4 | Leopardus pardalis | -82.858061 | 9.740033 | POINT (-82.85806 9.74003) |
# Valores únicos de la columna species
felidos_gdf['species'].unique()
array(['Leopardus pardalis', 'Panthera onca', 'Leopardus wiedii',
'Puma concolor', 'Puma yagouaroundi', 'Leopardus tigrinus'],
dtype=object)
# Conexión al servicio WFS del Sinac
wfs_url = 'http://geos1pne.sirefor.go.cr/wfs'
wfs_version = '1.1.0'
wfs = WebFeatureService(url=wfs_url, version=wfs_version)
# Obtener la capa de áreas de conservación
capa = 'PNE:areas_conservacion'
respuesta = wfs.getfeature(typename=capa, outputFormat='application/json')
# Leer la respuesta en un GeoDataFrame
areas_conservacion_gdf = gpd.read_file(BytesIO(respuesta.read()))
# Reducción de columnas
areas_conservacion_gdf = areas_conservacion_gdf[['id', 'siglas_ac', 'nombre_ac', 'geometry']]
# Definir un índice
areas_conservacion_gdf.set_index('id', inplace=True)
# Reproyectar a WGS84
areas_conservacion_wgs84_gdf = areas_conservacion_gdf.to_crs(epsg=4326)
# Mostrar el GeoDataFrame
areas_conservacion_wgs84_gdf
| siglas_ac | nombre_ac | geometry | |
|---|---|---|---|
| id | |||
| areas_conservacion.1 | ACOSA | Osa | POLYGON ((-82.90321 8.73373, -82.90317 8.73357... |
| areas_conservacion.2 | ACT | Tempisque | POLYGON ((-85.43351 10.44009, -85.43309 10.439... |
| areas_conservacion.3 | ACG | Guanacaste | POLYGON ((-85.35777 11.12637, -85.22911 11.063... |
| areas_conservacion.4 | ACTo | Tortuguero | POLYGON ((-83.69448 10.93883, -83.69312 10.938... |
| areas_conservacion.5 | ACLAP | La Amistad Pacifico | POLYGON ((-83.49122 9.61393, -83.44427 9.61357... |
| areas_conservacion.6 | ACOPAC | Pacifico Central | POLYGON ((-84.82622 10.33726, -84.82587 10.337... |
| areas_conservacion.7 | ACC | Central | POLYGON ((-84.01143 10.44435, -84.01105 10.444... |
| areas_conservacion.8 | ACAHN | Huetar Norte | MULTIPOLYGON (((-84.08375 10.29133, -84.08375 ... |
| areas_conservacion.9 | ACAT | Arenal Tempisque | POLYGON ((-85.09671 10.78235, -85.09685 10.746... |
| areas_conservacion.10 | ACLAC | La Amistad Caribe | MULTIPOLYGON (((-83.5426 9.97478, -83.54267 9.... |
Análisis#
# Unión espacial
felidos_ac_gdf = felidos_gdf.sjoin(
areas_conservacion_wgs84_gdf,
predicate='intersects'
)
felidos_ac_gdf.head()
| species | decimalLongitude | decimalLatitude | geometry | id | siglas_ac | nombre_ac | |
|---|---|---|---|---|---|---|---|
| 0 | Leopardus pardalis | -83.530469 | 10.595040 | POINT (-83.53047 10.59504) | areas_conservacion.4 | ACTo | Tortuguero |
| 1 | Panthera onca | -84.000000 | 9.950000 | POINT (-84 9.95) | areas_conservacion.7 | ACC | Central |
| 2 | Panthera onca | -85.050000 | 10.968080 | POINT (-85.05 10.96808) | areas_conservacion.8 | ACAHN | Huetar Norte |
| 3 | Leopardus wiedii | -85.624341 | 10.370696 | POINT (-85.62434 10.3707) | areas_conservacion.2 | ACT | Tempisque |
| 4 | Leopardus pardalis | -82.858061 | 9.740033 | POINT (-82.85806 9.74003) | areas_conservacion.10 | ACLAC | La Amistad Caribe |
# Conteo de especies en cada ASP
conteo_especies_por_ac = felidos_ac_gdf.groupby("id").species.nunique()
# Convertir la serie a dataframe
conteo_especies_por_ac = conteo_especies_por_ac.reset_index()
# Definir un índice
conteo_especies_por_ac.set_index('id', inplace=True)
# Cambio de nombre de columna
conteo_especies_por_ac.rename(columns = {'species': 'especies_felidos'}, inplace = True)
# Despliegue de áreas de conservación por cantidad de especies
conteo_especies_por_ac.sort_values(by="especies_felidos", ascending=False)
| especies_felidos | |
|---|---|
| id | |
| areas_conservacion.7 | 6 |
| areas_conservacion.1 | 5 |
| areas_conservacion.9 | 5 |
| areas_conservacion.10 | 4 |
| areas_conservacion.2 | 4 |
| areas_conservacion.3 | 4 |
| areas_conservacion.6 | 4 |
| areas_conservacion.8 | 4 |
| areas_conservacion.5 | 3 |
| areas_conservacion.4 | 2 |
# Join para agregar la columna con el conteo a la capa de áreas de conservación
areas_conservacion_wgs84_gdf = areas_conservacion_wgs84_gdf.join(conteo_especies_por_ac)
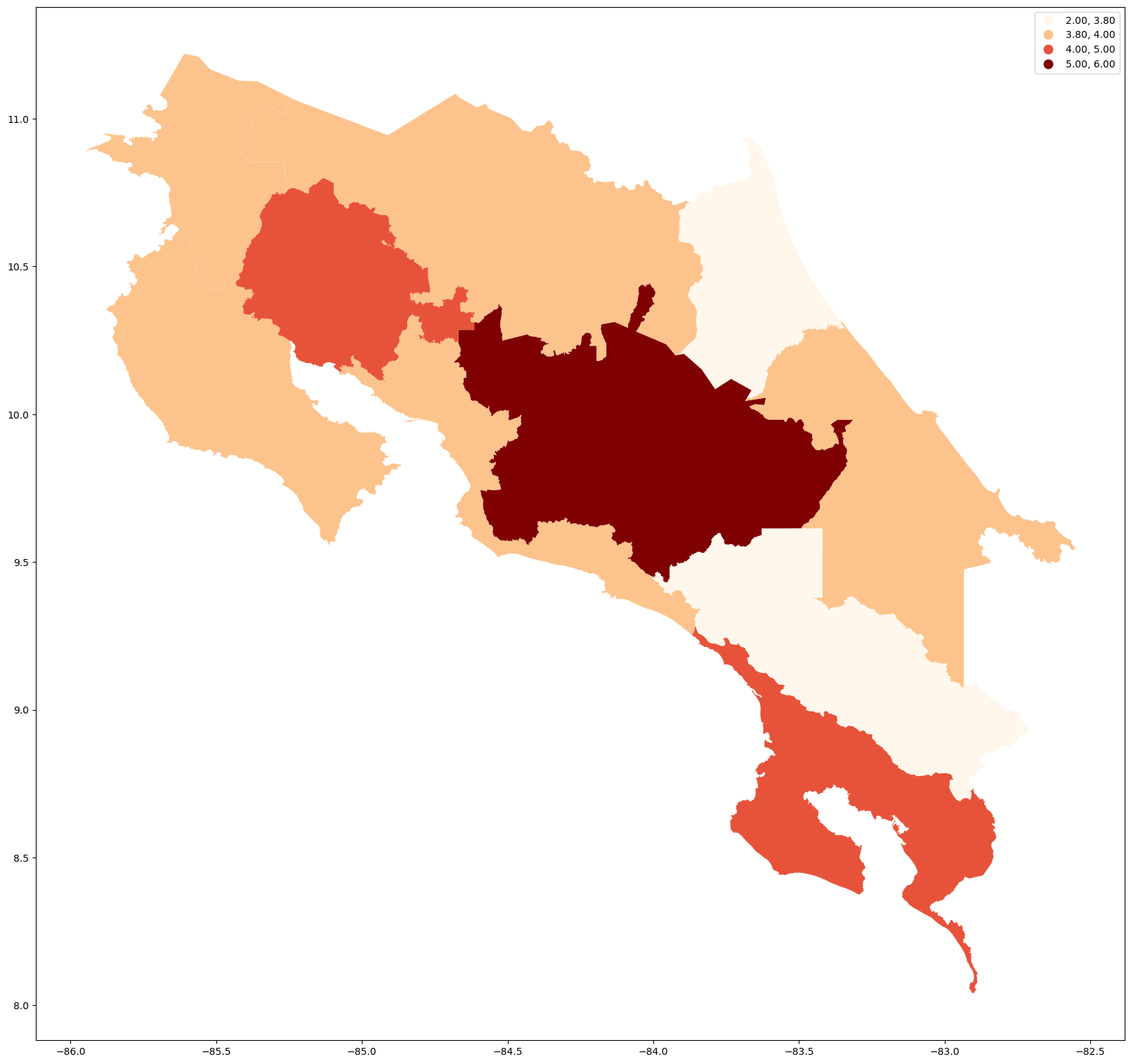
areas_conservacion_wgs84_gdf.sort_values(by='especies_felidos', ascending=False)
| siglas_ac | nombre_ac | geometry | especies_felidos | |
|---|---|---|---|---|
| id | ||||
| areas_conservacion.7 | ACC | Central | POLYGON ((-84.01143 10.44435, -84.01105 10.444... | 6 |
| areas_conservacion.1 | ACOSA | Osa | POLYGON ((-82.90321 8.73373, -82.90317 8.73357... | 5 |
| areas_conservacion.9 | ACAT | Arenal Tempisque | POLYGON ((-85.09671 10.78235, -85.09685 10.746... | 5 |
| areas_conservacion.2 | ACT | Tempisque | POLYGON ((-85.43351 10.44009, -85.43309 10.439... | 4 |
| areas_conservacion.3 | ACG | Guanacaste | POLYGON ((-85.35777 11.12637, -85.22911 11.063... | 4 |
| areas_conservacion.6 | ACOPAC | Pacifico Central | POLYGON ((-84.82622 10.33726, -84.82587 10.337... | 4 |
| areas_conservacion.8 | ACAHN | Huetar Norte | MULTIPOLYGON (((-84.08375 10.29133, -84.08375 ... | 4 |
| areas_conservacion.10 | ACLAC | La Amistad Caribe | MULTIPOLYGON (((-83.5426 9.97478, -83.54267 9.... | 4 |
| areas_conservacion.5 | ACLAP | La Amistad Pacifico | POLYGON ((-83.49122 9.61393, -83.44427 9.61357... | 3 |
| areas_conservacion.4 | ACTo | Tortuguero | POLYGON ((-83.69448 10.93883, -83.69312 10.938... | 2 |
# Mapa estático
areas_conservacion_wgs84_gdf.plot(
column="especies_felidos",
legend=True,
cmap='OrRd',
scheme='quantiles',
figsize=(20, 20)
)
/home/mfvargas/miniconda3/envs/gf0657-2024-ii/lib/python3.12/site-packages/mapclassify/classifiers.py:1653: UserWarning: Not enough unique values in array to form 5 classes. Setting k to 4.
self.bins = quantile(y, k=k)
<Axes: >
# Crear el mapa interactivo con las áreas de conservación
m = areas_conservacion_wgs84_gdf.explore(
column='especies_felidos',
name='Riqueza de especies de félidos',
cmap='OrRd',
tooltip=['nombre_ac', 'siglas_ac', 'especies_felidos'],
legend=True,
legend_kwds={
'caption': "Riqueza de especies de félidos en áreas de conservación",
'orientation': "horizontal"
},
)
# Añadir los registros de félidos al mapa
felidos_gdf.explore(
m=m, # se usa el mapa que se creó en la instrucción anterior
name='Registros de presencia de félidos',
marker_type='circle',
marker_kwds={'radius': 20, 'color': 'black'},
tooltip=['species'],
popup=True
)
# Agregar un control de capas al mapa
folium.LayerControl().add_to(m)
# Mostrar el mapa interactivo
m
Make this Notebook Trusted to load map: File -> Trust Notebook
Ejercicios#
Programe un mapa interactivo de coropletas que muestre la cantidad de aeródromos en los cantones de Costa Rica. Busque las fuentes de datos en el SNIT.


